熱線:021-66110810,66110819
手機:13564362870


熱線:021-66110810,66110819
手機:13564362870
3.2. Acd_DPN7 3SP-CoA 脫亞磺酰酶與脫氫酶的總體序列和折疊相似性
3SP-CoA 脫亞磺酰酶 Acd_DPN7 與酰基輔酶 A 脫氫酶具有高度的結構相似性,并且每個亞基含有一個 FAD。因此,它與任何先前報道的脫亞磺酰酶在結構上無關。Acd_DPN7 單體 A 與幾種其他酰基輔酶 A 脫氫酶坐標的平均 r.m.s.d. 約為 1.5 ?。Acd_DPN7 已經進化為脫亞磺酰酶,同時保留了酰基輔酶 A 脫氫酶超家族中常見的 38% 的氨基酸殘基。對于酰基輔酶 A 脫氫酶具有脫氫酶活性,在 246 位或 366 位擁有催化堿基(Glu 或 Asp 殘基)是必不可少的。在 Acd_DPN7 中,這兩個位置都沒有發現谷氨酸:在 246 位發現一個谷氨酰胺殘基,在 366 位發現一個甘氨酸殘基。在 246 位或 366 位缺少 Glu 殘基解釋了為什么 Acd_DPN7 和其他最近鑒定的脫亞磺酰酶無法催化任何測試的酰基輔酶 A 底物的脫氫。在對應于 Acd_DPN7 中 Gln246 的酰基輔酶 A 脫氫酶中最常出現的殘是 Gly(七次)、Thr(四次)、Ala(三次)和 Glu(兩次)。在對應于 Acd_DPN7 中 Gly366 的位置,發現最常出現的殘基是 Glu(14 次)和 Ala(兩次)。Gly366Glu 和 Gly366Ala 突變都可以解釋為單核苷酸交換的結果,即從谷氨酸的 DNA 序列 GAA/GAG (Glu) 變為 GGA (Gly) 或 GCA/GCG (Ala)。
3.3. 活性位點的鑒定和表征
來自 Desulfococcus multivorans 的戊二酰輔酶 A 脫氫酶與戊二酰輔酶 A 復合物的結構,以及來自豬肝線粒體的中鏈酰基輔酶 A 脫氫酶與辛酰輔酶 A 復合物的結構,與 Acd_DPN7 3SP-CoA 脫亞磺酰酶結構的疊加表明,圍繞殘基 Arg84、Asp88、Lys118-Ile121、Tyr243-Gln246 和 FAD 異咯嗪環的空腔可能是 Acd_DPN7 3SP-CoA 脫亞磺酰酶的反應中心。為了驗證這一假設,Acd_DPN7 在存在琥珀酰輔酶 A 的情況下結晶,琥珀酰輔酶 A 代表了 3-亞磺酰丙酰輔酶 A (3SP-CoA) 的結構類似物。琥珀酰部分的 C 原子編號為 1-4,位置 4 對應于末端羧酸鹽。在 3SP-CoA 中,位置 4 對應于末端亞磺酰基(圖 3)。共結晶,隨后浸泡琥珀酰輔酶 A,導致琥珀酰輔酶 A 水解。正如 Wischgoll 等人報道的那樣,抑制二羧酸輔酶 A 硫酯的水解是共結晶的主要挑戰,因為從大多數數據集計算的電子密度僅包含輔酶 A。
在 Acd_DPN7 單體 A、B、D、E 和 F 的活性位點中,僅基于電子密度圖可以觀察到輔酶 A 基團(圖 4 )。對于單體 C,沒有觀察到與輔酶 A 基團相關的電子密度,但在空腔深處模擬了一個琥珀酰基團。在不含底物的情況下計算的 Fo-Fc 差值傅里葉 OMIT 圖,在 3.0σ 輪廓水平(圖 4 ),標記了腺苷基團、核糖醇基團、三個磷酰基團、泛酸部分的羰基和輔酶 A S 原子的位置,從而指導了整個輔酶 A 基團的定位。由 PHENIX 計算的結合在單體 A、B、D、E 和 F 中的輔酶 A 基團的實空間相關系數分別為 0.7、0.81、0.73、0.77 和 0.75。使用 OVERLAPMAP 計算的與 2Fo-Fc 圖的相關因子分別為 0.50(0.66)、0.57(0.70)、0.54(0.63)、0.57(0.69) 和 0.59(0.75)。括號中的值是使用 2Fo-Fc 特征增強圖計算的相關因子(圖 4 )。
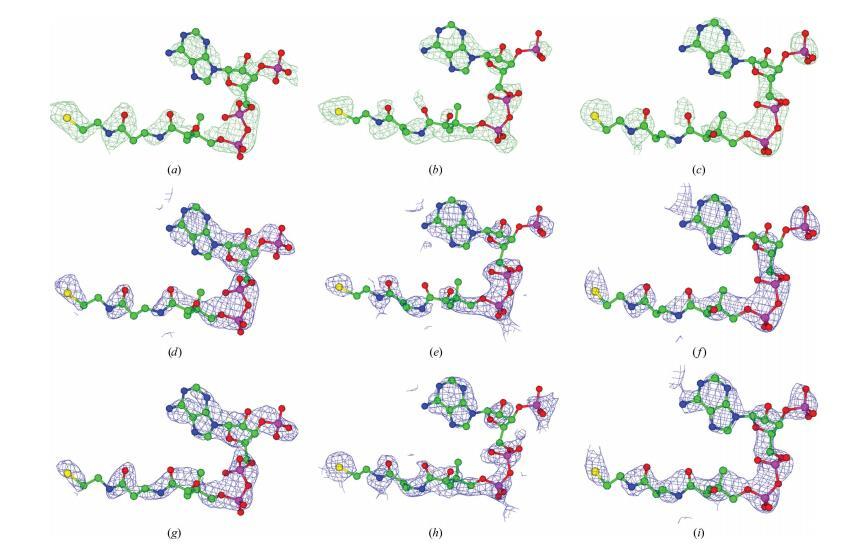
以球棍模型展示的單體B(a、d、g)、D(b、e、h)和F(c、f、i)的輔酶A基團(原子顏色編碼如下:碳,綠色;氧,紅色;磷,品紅色;氮,藍色;硫,黃色)分別擬合于:(上排)FoFc差值傅里葉OMIT圖(綠色網格;等值線水平3.0,未含底物計算);(中排)2FoFc差值傅里葉圖(藍色網格;等值線水平1.0,使用phenix軟件計算, Adams 等,2010);(下排)2FoFc差值傅里葉圖(藍色網格;等值線水平2.0,采用phenix的‘特征增強’選項)。
我們精修了五個輔酶 A 基團的占有率,以獲得接近周圍蛋白質殘基觀察到的 B 因子值。單體 A、B、D、E 和 F 的輔酶 A 基團的最終占有率值分別為 0.43(57 ?2)、0.48(61 ?2)、0.57(60 ?2)、0.53(67 ?2) 和 0.59(72 ?2)(括號中給出 B 因子值)。基于 R 因子的完整結構(包括精修的輔酶 A 基團)的 Cruickshank DPI 坐標誤差為 0.3 ?。在脫輔基酶的晶體結構中沒有觀察到讓人聯想到 PEG 存在的電子密度,PEG 用于天然酶和復合物的結晶和冷凍保護,因此也排除了其在復合物結構活性位點中的存在。此外,復合物的結晶或冷凍保護沒有使用甘油,因為其在天然酶的活性位點中 FAD 異咯嗪環上方被觀察到。在復合物結構活性位點的深處,在 2F-Fc 傅里葉圖中僅觀察到少數水分子,輪廓水平為 1.0σ。輔酶 A 基團在 Acd_DPN7 空腔內的定位在單體之間是一致的,從而允許鑒定酶的活性位點。
輔酶 A 的腺苷基團與 Asn244 和 Arg247 的側鏈處于氫鍵距離。特別是,Asn244 的 OD1 原子距離腺苷基團的 N6A 原子 2.9 ?,而 Asn244 的 ND2 原子距離腺苷基團的 N1A 原子 3.2 ?(報告的值是針對單體 D 的,其余單體的值相似)。Arg247 參與輔酶 A 部分的精確定位,正如在我們的結構中觀察到的那樣。在酰基輔酶 A 脫氫酶的其他結構中,對應于 Arg247 的精氨酸殘基是嚴格保守的。在大多數情況下,它們與輔酶 A 酰胺基團的兩個羰基 O 原子相互作用。在中鏈酰基輔酶 A 脫氫酶 (MCADs) 中,有兩個 Arg 殘基與輔酶 A 部分相互作用,分別是 Arg324(與輔酶 A 腺嘌呤環相互作用)和 Arg256(與羰基 O 原子相互作用)。在 Acd_DPN7 中,沒有 MCAD 中 Arg324 的等效物,而 Arg247(對應于 MCAD 中的 Arg256)靠近輔酶 A 的羰基 O 原子和腺嘌呤環。Arg247 可能在識別腺苷基團中起作用。在人類戊二酰輔酶 A 脫氫酶中發現了 Arg250Trp 突變(對應于 Acd_DPN7 中的 Arg247)是致病性的。
 相關新聞
相關新聞