熱線:021-66110810,66110819
手機:13564362870


熱線:021-66110810,66110819
手機:13564362870
輔酶 A 的三個磷酰基距離 Lys384 3.1 ?,并束縛著一個水分子網絡。幾個疏水殘基(Phe236、Met240 和 Ile316)定義了底物結合口袋的入口。輔酶 A 泛酸部分的 NH 基團與 Ser130 主鏈的羰基在 2.7 ? 處形成氫鍵。Arg247 側鏈位于輔酶 A 泛酸部分內的羰基之間。輔酶 A S 原子位于平面黃素基團上方,距離其 3.1 ?,距離 Tyr243 OH 基團 3.4 ?。Tyr243 的存在增加了空腔的極性,相比之下,在其他 Acds 中,相應位置通常發現苯丙氨酸或亮氨酸。
脫輔基形式和類似物復合物晶體的晶胞內容物不同,脫輔基形式中有一個二聚體,復合物結構中有三個二聚體。兩種晶體都屬于相同的空間群 (P21212),并具有相同的 Matthews 系數。它們的晶胞尺寸通過 (0,1,0;3,0,0;0,0,1) 變換矩陣相關聯。在晶格中,這些變化通常是由于冷凍保護、冷卻和/或浸泡化合物的差異而發生的,這些化合物結合到晶體中并可能扭曲晶格,導致晶胞參數和空間群發生變化。在這種情況下,添加新鮮的琥珀酰輔酶 A 和從冷凍條件中省略甘油可能導致浸泡晶體的晶胞重排。Acd_DPN7 單體 A(殘基 2-392)在脫輔基形式和與輔酶 A 復合物的單體疊加顯示,僅外部環中的殘基 Gly143、Asp173 和 Gly190 的 r.m.s. 偏差(>1 ?)。Acd_DPN7 單體 A 和與輔酶 A 復合物的每個單體,在殘基 2-392 的 C 原子上計算的 r.m.s. 偏差 <0.5 ?。
3.4. Acd_DPN7 3SP-CoA 脫亞磺酰酶中 3SP-CoA 的建模
來自 D. multivorans 的戊二酰輔酶 A 脫氫酶與戊二酰輔酶 A 復合物的結構,以及來自豬肝線粒體的中鏈酰基輔酶 A 脫氫酶與辛酰輔酶 A 復合物的結構,與含有輔酶 A 基團的 Acd_DPN7 3SP-CoA 脫亞磺酰酶結構的疊加證實,戊二酰輔酶 A 和辛酰輔酶 A 與各自脫氫酶的結合模式與輔酶 A 在 Acd_DPN7 空腔內的結合模式相同。
為了研究活性位點中哪些殘基可能與底物的末端亞磺酰基相互作用,基于結合的輔酶 A 部分的結構對 3SP-CoA 進行了建模(圖 5a)。然后對模型進行一輪分子動力學 (phenix.dynamics),使用默認設置以消除模型偏差并獲得正確的幾何形狀。
這清楚地顯示了丙酰基(C 原子位置 1-3;圖 3)的定位,亞磺酰基的 S 原子(位置 4)距離 Arg84、Asp88 和 Gln246 的側鏈在 3.5 ? 以內,位于 FAD 異咯嗪環平面的上方(圖 5a 和 5b)。一個水分子網絡可以容納在亞磺酰基的氫鍵距離內(圖 5a)。
在這個模型中,3SP-CoA 硫酯鍵的羰基 O 原子既與 FAD 核糖醇側鏈的 2'-OH 形成氫鍵,也與 Gly366 的主鏈 NH 形成氫鍵。通常,在酰基輔酶 A 脫氫酶中,底物硫酯鍵的羰基 O 原子既與 FAD 核糖醇側鏈的 2'-OH 形成氫鍵,也與 Glu376 的主鏈 NH 形成氫鍵。這些相互作用對于底物羰基的取向和極化很重要。在脫氫酶的情況下,底物羰基的極化對于降低底物 α-碳的 pKa 值是必要的,這是脫氫反應所必需的。這種極化效應在 3SP-CoA 的脫亞磺酰反應中是否也很重要,仍有待理解。
因此,Arg84、Asp88 和 Gln246 的側鏈以及 FAD 的異咯嗪環定義了脫亞磺酰反應的酶促環境。3SP-CoA S4 與 FAD 異咯嗪環的距離將 >3.8 ?。
3.5. 突變酶 Acd_DPN7_R84K 和 Acd_DPN7_Q246E
Acd_DPN7 3SP-CoA 脫亞磺酰酶活性位點內 3SP-CoA 的建模將亞磺酰基定位在靠近 Arg84 的位置,Arg84 NH2 與 3SP-CoA OS5 的距離為 3.2 ?(圖 5b)。殘基 Arg84 已被提議參與結合 3SP-CoA 的亞磺酰基,因為在戊二酰輔酶 A 脫氫酶中觀察到了類似的相互作用。在這些酶中,一個精氨酸殘基參與結合末端羧基。為了研究該殘基對脫亞磺酰反應是否必不可少,將 Arg84 突變為賴氨酸,產生 Acd_DPN7_R84K 變體。野生型 Acd_DPN7 和 Acd_DPN7_R84K 的尺寸排阻色譜表明,Acd_DPN7_R84K 被純化為完整的四聚體,因此是正確折疊的蛋白質。Acd_DPN7_R84K 的吸收光譜與 Schürmann 等人發表的 Acd_DPN7 光譜相同。這表明所有 FAD 結合位點都已飽和,并且四聚體正確折疊。將 Arg84 突變為 Lys 導致活性完全喪失(表 3),表明 Arg84 具有重要作用。
亞磺酸比其對應的羧酸酸性更強,因此在生理 pH 下預計會去質子化。Arg84 的胍基 pKa 為 12.5,預計在生理 pH 下帶正電荷,從而穩定帶負電荷的亞磺酰基。考慮到氨基和胍基的 pKa 相似(Lys 和 Arg 分別為 11 和 12.5),預計兩個側鏈在生理 pH 下都會質子化,并且都應該能夠穩定帶負電荷的亞磺酰基。Arg84 將帶有一個在胍基上離域的正電荷,而賴氨酸將在 NZ 原子上帶有一個非常局域化的正電荷。因此,電荷的穩定可能只是 Arg84 側鏈作用的一個方面。此外,胍陽離子是強氫鍵供體。
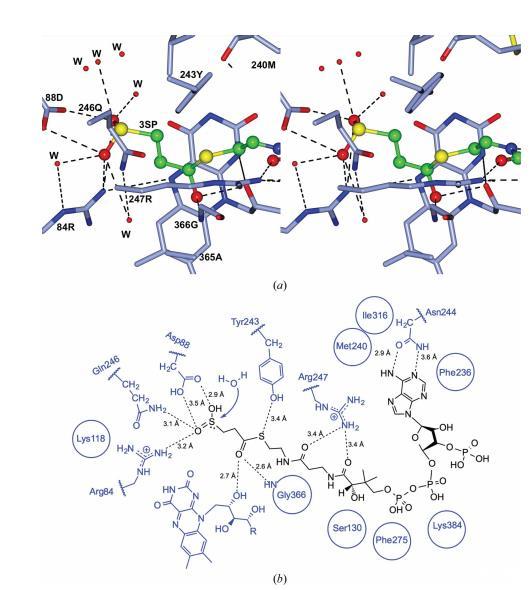
圖5 顯示了通過分子動力學最小化處理后,A. mimigardefordensis來源的AcdDPN7(3SP-CoA脫硫酶)活性位點中3SP-CoA基團的定位視圖。3SP-CoA的定位基于3SP-CoA脫硫酶腔內CoA的晶體學精修位置。(a)腔深端立體視圖。3SP-CoA以球棍模型表示,原子顏色編碼如下:碳原子綠色、氧原子紅色、氮原子藍色、硫原子黃色。蛋白質以青色粗鍵表示,其中氧原子為紅色、氮原子為藍色。水分子(W)以紅色球體表示。(b)整個結合位點的二維表示。
在模型中,Gln246 NE2 與 3SP-CoA OS5 的距離為 3.1 ?(圖 5b),并且先前假設 Gln246 的存在是 Acd_DPN7 無法催化酰基輔酶 A 脫氫酶反應的原因。因此,產生了 Acd_DPN7_Q246E 變體。該變體仍然能夠作為脫亞磺酰酶(表 3),但不能利用任何測試的酰基輔酶 A 脫氫酶底物(丙酰輔酶 A、丁酰輔酶 A、異丁酰輔酶 A、戊酰輔酶 A、異戊酰輔酶 A、琥珀酰輔酶 A、戊二酰輔酶 A 和 3SP-CoA)(結果未顯示)。因此,Acd_DPN7_Q246E 突變體在谷氨酰胺突變為谷氨酸后并未獲得酰基輔酶 A 脫氫酶活性。然而,在 Acd_DPN7_Q246E 突變體中,取代對脫亞磺酰速率有輕微影響(表 3)。Km 從野生型到突變體增加了約 46%,而 kcat 值增加了約 25%。底物結合似乎部分受損,可能是由于新添加的帶負電荷的羧基(谷氨酰胺突變為谷氨酸),這可能會排斥帶負電荷的 3-亞磺酰丙酰基的進入。反應速率可能因空腔帶負電荷更多而增強,這將有助于在脫亞磺酰后去除 HSO3 基團。
 相關新聞
相關新聞